近日,农学院张文利课题组在 “Nucleic Acids Research”上发表了题为“The roles of DNA methylation on pH dependent i-motifs (iMs) formation in rice”的研究论文。该研究是在前期的研究基础之上(Nucleic Acids Res 2022, 50:3226-3238),再次利用iM-IP-seq方法,从全基因组水平较为系统地解析了水稻基因组DNA甲基化修饰水平和pH条件影响iMs结构形成的复杂关系。
在人和动植物基因组中,i-motifs(iMs)是一类普遍存在且十分重要的非B型DNA二级结构,其在维持基因组结构稳定性、DNA复制、基因转录和翻译等多种生物学过程中发挥着重要的调控作用。目前,多数研究发现,体外iMs的形成严格依赖于酸性pH环境,且DNA甲基化水平也可影响基因组部分位点的iMs形成。最新研究结果表明,在正常生理条件下也可形成iMs结构,但其形成的机理不清。迄今为止,不同pH条件如何影响iM的形成及其分布还鲜有报道。特别是, DNA甲基化修饰水平和pH条件如何影响iMs的形成仍缺乏系统研究。该问题的解决将有助于人们理解在细胞正常生理条件下形成iMs的可能分子机制。
该研究首先利用iM-IP-seq方法,绘制了pH 5.5及pH 7.0 条件下水稻基因组iMs分布图谱。分别鉴定了33,455个pH 5.5和55,062个pH 7.0 特异的iMs位点。基因组分布结果显示,pH 7.0 特异的iMs位点主要分布于5’UTRs、基因间区、基因下游区域及内含子区域;相比之下,pH 5.5特异的iMs位点主要分布在外显子区域。
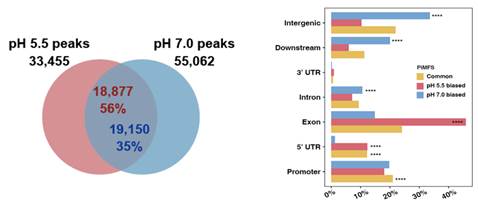
图1. pH 5.5及pH 7.0 条件下iMs鉴定及其在亚基因组分布
DNA甲基化水平的分析结果表明,pH 7.0 特异的iMs呈现出较高的甲基化修饰水平,与pH 5.5特异的iMs甲基化修饰水平明显相反。这一结果暗示,DNA甲基化水平可能是影响不同pH条件下iMs形成的关键因素之一。
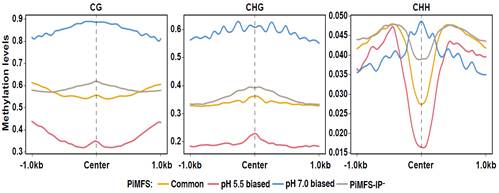
图2. pH 7.0和 pH 5.5特异的iMs的甲基化修饰水平比较
进一步结合全基因组高低DNA甲基化的iM-IP-seq,发现DNA甲基化水平降低对pH 7.0条件下iMs形成的影响要大于pH 5.5的;而DNA甲基化水平升高则更不利于pH 5.5条件下iMs的形成。重要的是,我们发现只有一定程度的甲基化修饰水平有于不同pH依赖的iMs的形成,过高或过低的甲基化水平均不利于iMs的形成。
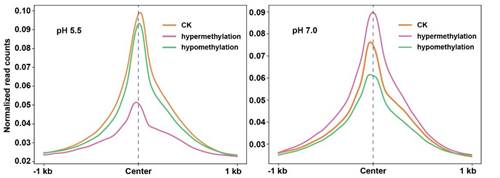
图3 高低DNA甲基化对pH 7.0和pH 5.5条件下iM形成的影响
最后,分析结果表明,在pH5.5和7.0条件下,CG-hypo-DMRs和CHH-hyper-DMRs单独或与CG/CHG-hyper-DMRs协同的作用,共同影响iMs的形成。
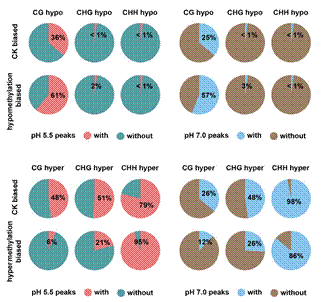
图4. pH协同高/低DNA甲基化影响iM的形成
总之,本研究首次在全基因组水平,较为系统地研究了DNA甲基化和pH条件影响iMs形成的复杂关系,一方面初步阐明了细胞正常生理条件下形成iMs的可能分子机制,同时促进了水稻iMs生物学功能的研究及其潜在的应用价值。
南京农业大学钟山青年研究员冯逸龙和陶申童博士、已毕业硕士马星和在读硕士生杨滢为论文共同第一作者,张文利教授为通讯作者。南京农业大学程雪娇副教授以及博士留学生Asgar Ahmed,扬州大学龚志云教授等也参与了该研究。该研究得到了国家重点研发项目(2022YFA1303400)和国家自然科学基金(32201782, 32070561, 及U20A2030)等课题的资助;同时感谢南京农业大学作物遗传与种质创新利用全国重点实验室及校生物信息学研究中心为本研究提供的平台支撑。
本文链接:https://doi.org/10.1093/nar/gkad1245